北京基因组所(国家生物信息中心)合作开发单细胞转录组数据仿真软件SimCH
近十多年来,单细胞RNA测序技术(scRNA-seq)不断发展和普及,同时数以千计的生物信息学/计算生物学工具被开发出来,以解决诸如数据获取(包括量化、比对、拼接等)、数据清洗(包括质控、过滤、归一化、整合等)、细胞分配(包括聚类、排序、分类等)、基因识别(包括基因网络、差异表达、标记基因等)等scRNA-seq数据处理和分析步骤。尽管其中大部分软件经过同行评议且在其论文中也彰显了优点,但它们自己的比较可能存在偏好(bias),其可靠性尚未得到系统性检测(benchmark)。
近年来出现了一些分别针对差异表达、聚类、插补、轨迹推断、基因调控网络推断等分析的第三方系统性检测,通常使用大量的实验数据和仿真数据作为基准参考,来评估多个同类型方法在各项指标上的性能优劣。虽然实验数据因其真实性较好,通常作为工具评估的“金标准”,但实验数据的获取和适合性也存在问题。相比较而言,仿真数据具有能快速产生、经济、扩展性好等优点,可以作为工具评估的“银标准”。近日,中科院北京基因组所(国家生物信息中心)张治华研究团队与扬州大学孙磊副教授联合在生物信息学著名期刊Briefings in Bioinformatics发表了题为SimCH: simulation of single cell RNA sequencing data by modeling cellular heterogeneity at gene expression level的研究型论文,推出了一款用于产生scRNA-seq仿真数据的软件--SimCH。SimCH是一种半参数化(semi-parametric)的生成模型,它基于实验数据估计统计学模型参数,并提供给用户修改参数(如细胞数量、基因数量、测序深度、是否零膨胀)的接口,主要模式有SimCH-flex、SimCH-fit、SimCH-copula及扩展模式SimCH-ext。SimCH能通过高斯Copula模型在仿真数据中保留实验数据的基因共表达信息。通过配置合适的参数,SimCH产生的仿真数据能够很好地拟合同质性/异质性+UMI/非UMI技术的scRNA-seq实验数据。实验结果表明SimCH的整体性能优于现流行的仿真工具Splat和2021年推出的仿真工具scDesign2。
SimCH可应用于评估诸如细胞聚类、差异表达分析、轨迹推断、插补等不同类型的计算方法性能。研究人员还发现,基因共表达信息与数据样本的异质性密切相关。SimCH能够有力支持scRNA-seq计算方法的系统性检测,助力单细胞组学研究。
孙磊副教授为本文第一作者,孙磊和张治华研究员为共同通讯作者,硕士研究生王公铭参与了软件开发。项目得到国家自然科学基金的资助。
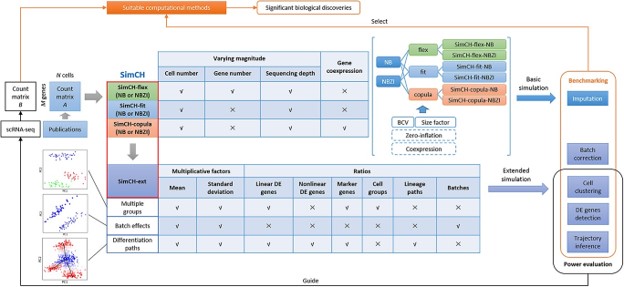
SIMCH运行框架图






