国家生物信息中心开发针对单细胞RNA测序数据的剪接分析新工具SCSES
RNA可变剪接是基因转录后调控的重要过程,其中蕴含着丰富的转录本结构和功能多样性信息,是细胞转录异质性的重要来源。尽管单细胞RNA测序技术已被广泛应用于研究细胞异质性,但由于测序深度低、数据噪音和缺失率高等问题,在单细胞尺度精确量化可变剪接事件仍存在较大挑战。
10月27日,国家生物信息中心计算生物学部刘肇祺研究团队在Nature Communications杂志在线发表了题为“Deciphering splicing heterogeneity at single-cell resolution by SCSES”的研究论文。提出一款名为SCSES的单细胞可变剪接事件识别与定量推断算法。该方法能够准确还原单细胞分辨率水平可变剪接的动态变化,为理解细胞类型与状态异质性提供了新的分析视角。
SCSES算法基于复杂网络信息传播模型,创新性地融合了细胞剪接相似性和剪接事件相似性等多维信息,实现对单个细胞剪接状态的推断。该算法利用RNA绑定蛋白基因表达谱、原始junction测序读段和原始PSI矩阵构建了细胞剪接相似性网络,并整合剪接事件序列特征及剪接调控关系构建了剪接事件相似性网络。最后,面对数据中存在的四种典型缺失值情境,SCSES基于不同的相似性网络设计了三种针对性的缺失值补全策略,实现了单细胞内剪接强度的准确刻画。
研究团队通过模拟数据和真实生物学数据系统评估了SCSES与当前已有可变剪接分析方法的性能差异。结果显示,SCSES在剪接事件强度恢复准确性和差异剪接事件识别方面均优于现有方法。同时,在多种干细胞和胚胎发育数据集中,SCSES在细胞亚群划分与发育轨迹重建中也展现出良好的生物学一致性。
实际应用中,SCSES在多种重要生物学场景中展现出对细胞异质性更高的分辨能力。在多发性骨髓瘤耐药性研究中,SCSES在初诊患者样本中识别出了具有潜在硼替佐米耐药特征的细胞亚群,这一发现无法通过传统基因表达分析获得;在胚胎发育研究中,SCSES精细解析了中内胚层向内胚层转化过程中的剪接动态,揭示关键剪接事件可能参与调控细胞分化进程。此外,SCSES也具有在基于微流控技术的数据中进行细胞异质性分析的能力。在基于inDrop平台的造血干细胞分化数据集中,SCSES成功解析了单核细胞亚群间的剪接异质性。以上结果表明,SCSES是一个具备多场景适应能力的单细胞剪接分析工具,可有效应用于不同生物背景、物种及测序平台的剪接分析,为肿瘤异质性、发育生物学及疾病机制研究提供了有力工具。
国家生物信息中心刘肇祺研究员为本文的通讯作者,博士后温潇和博士生吕萱为本文的共同第一作者。该研究得到了国家重点研发计划和国家自然科学基金等项目的资助。
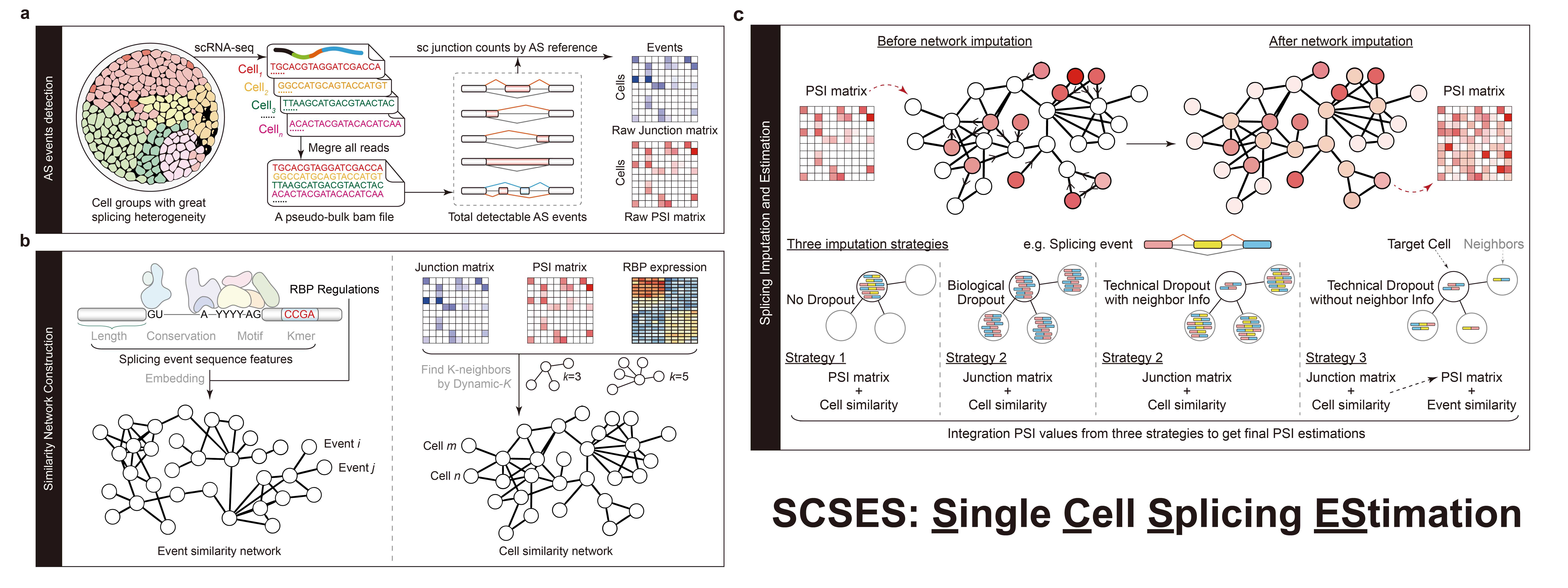
SCSES计算流程框架






