国家生物信息中心发布更新版全转录组关联研究知识库TWAS Atlas 2.0
近日,国家生物信息中心开发的全转录组关联研究知识库TWAS Atlas 2.0正式上线。该研究内容以“TWAS Atlas 2.0: an updated data resource for transcriptome-wide association studies”为题在国际学术期刊Nucleic Acids Research 在线发表。
全转录组关联研究(Transcriptome-wide association studies, TWAS)通过整合遗传变异与基因表达数据精准识别疾病或表型相关基因,在解析复杂致病机理方面发挥着重要作用。针对全球TWAS数据快速增长及数据缺乏整合等问题,国家生物信息中心于2023年正式推出TWAS Atlas知识库,旨在全面集成基因-性状关联信息,为人类复杂性状或疾病相关研究提供重要参考。自上线以来,TWAS Atlas在动脉粥样硬化性心血管疾病发生机制与疾病多组学关联研究等多项研究中得到广泛应用。
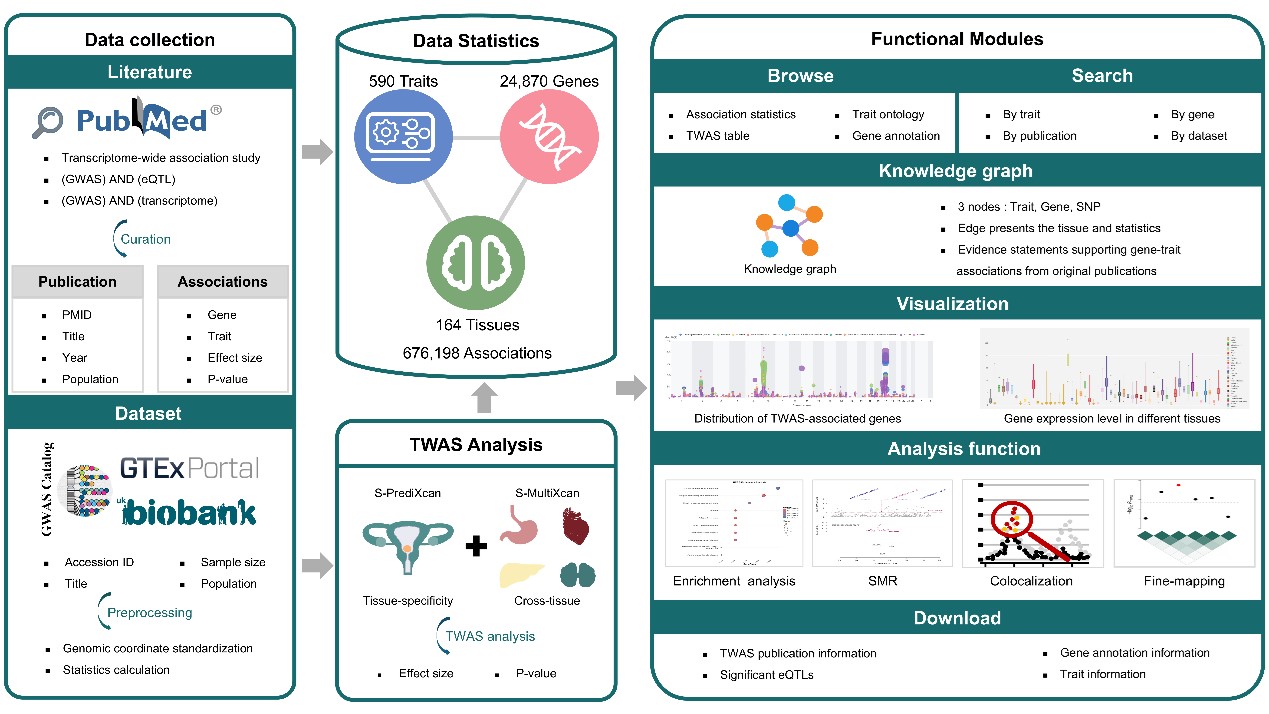
TWAS Atlas 2.0基于424篇TWAS文献的系统审编和171套GWAS数据集的自主分析,累计收录了676,198条基因-表型关联信息,涵盖590种人体性状与164种人体组织,并按照EFO、MeSH、UMLS等本体信息,建立了标准化的性状本体与分类体系,实现了TWAS性状名称、定义及类别的统一管理与精准标注。TWAS Atlas 2.0为用户提供多渠道的数据浏览、检索和下载功能,并对交互式SNP–基因–性状关联知识图谱进行了模块化升级。在实现多疾病、多组织、多组学关联调控关系解析及可视化的基础上,支持用户实时开展任意两种不同性状关联基因的重叠度对比分析,为研究人员探索复杂性状共性与特异性机制提供有力工具。此外,TWAS Atlas 2.0还提供功能富集、孟德尔随机化、共定位与精细匹配等多层次的分析结果展示,可与TWAS结果形成有效补充和交互验证,助力研究人员从多角度揭示基因、变异与性状间的调控规律。
国家生物信息中心硕士研究生高昊、工程师卜琮凡、中国医学科学院医学信息研究所郑思副研究员、国家生物信息中心博士研究生麦嘉琳为本文共同第一作者,中国医学科学院医学信息研究所李姣研究员、国家生物信息中心高级工程师曾瀞瑶和肖景发研究员为共同通讯作者。本研究得到国家重点研发计划、中科院战略性先导科技专项、国家自然科学基金以及中科院青促会等项目资助。
TWAS Atlas 2.0工作流程






