国家生物信息中心合作系统性评估单血统与多血统多基因评分方法并发布一站式在线计算平台PGS-hub
多基因评分(Polygenic Score,PGS)通过整合全基因组范围内大量遗传变异的微小效应,刻画个体对复杂性状的遗传易感性,在精准医学和疾病风险分层中具有重要应用前景。然而,不同PGS方法在建模假设、预测性能、计算复杂度、连锁不平衡(LD)处理以及跨人群适用性等方面或存在显著差异,目前仍缺乏在统一框架下的系统性比较。
近日,国家生物信息中心与首都医科大学附属北京潞河医院、美国博德研究所合作在Nature Communications在线发表题为“Comprehensive benchmarking single and multi ancestry polygenic score methods with the PGS-hub platform”的研究论文。该研究从多维度对9种单血统与4种多血统主流PGS方法进行了系统评估,并构建了一个集成化、标准化的在线计算平台PGS-hub,方便研究者在线便捷使用。该研究为PGS方法的选择应用与跨人群推广提供了重要参考框架和技术支撑。
该研究基于UK Biobank数据,选取了19种复杂疾病和17个数量性状,在欧洲和非洲人群中,对13种代表性PGS方法(包括单血统方法、多血统方法及集成策略)进行了全面评测,系统比较了各方法在预测准确性、风险分层能力、计算效率以及LD参考面板样本量和所采用的SNP位点数量等因素对预测性能的影响。
研究结果显示,在单血统分析中,LDpred2在大多数性状中表现出较为稳定且优异的预测性能;在多血统场景下,该研究提出的多血统整合策略LDpred2-multi方法整体优于现有主流多血统方法(如PRS-CSx和X-Wing),并在预测效果与计算效率之间取得了更好平衡。此外,该研究还发现,当LD参考面板样本量增加至约1,000–5,000后,PGS的预测性能趋于稳定,为实际应用中LD资源配置提供了定量依据。
该研究通过构建PGS-hub在线计算平台,为解决PGS方法在实际应用中部署复杂、参数设置繁琐、性能缺乏统一框架比较等问题提供了方案。在线平台集成的主流PGS软件,内置多血统LD参考面板,支持用户上传GWAS汇总统计数据并一站式完成多方法并行计算与结果输出。PGS-hub采用标准化的数据处理流程和自动化计算框架,降低了PGS方法使用的技术门槛,有助于提升多基因风险评分研究的可重复性与可比性,为不同研究团队开展PGS分析和临床应用提供了便捷在线计算平台。
该成果是研究团队在PGS领域长期系统研究的重要延展。此前,团队已在冠心病等复杂疾病的遗传风险预测方面取得一系列进展,包括开发多基因评分跨人群迁移及遗传背景矫正方法(JACC,2019,doi: 10.1016/j.jacc.2020.06.024.),冠心病多性状及多种族关联信息系统整合的多基因风险评分构建新方法,显著提升了冠心病风险预测的跨人群适用性及与预测准确性(Nature Medicine,2023,doi: 10.1038/s41591-023-02429-x)。在上述研究基础上,本研究进一步从方法学层面对单血统与多血统PGS模型进行统一评测,并通过PGS-hub平台实现标准化集成与开放共享,以期推动多基因评分研究的方法开发比较与临床应用验证,服务疾病风险分层与主动健康。
国家生物信息中心王斐助理研究员、汪敏先研究员和首都医科大学附属北京潞河医院翟光耀主任是本文的共同通讯作者。国家生物信息中心-美国博德研究所联合培养博士生陈星宇、国家生物信息中心助理研究员王斐、工程师赵宏强为本文的共同第一作者。这项工作得到了中国科学院战略性先导研究计划、国家自然科学基金委的资助。
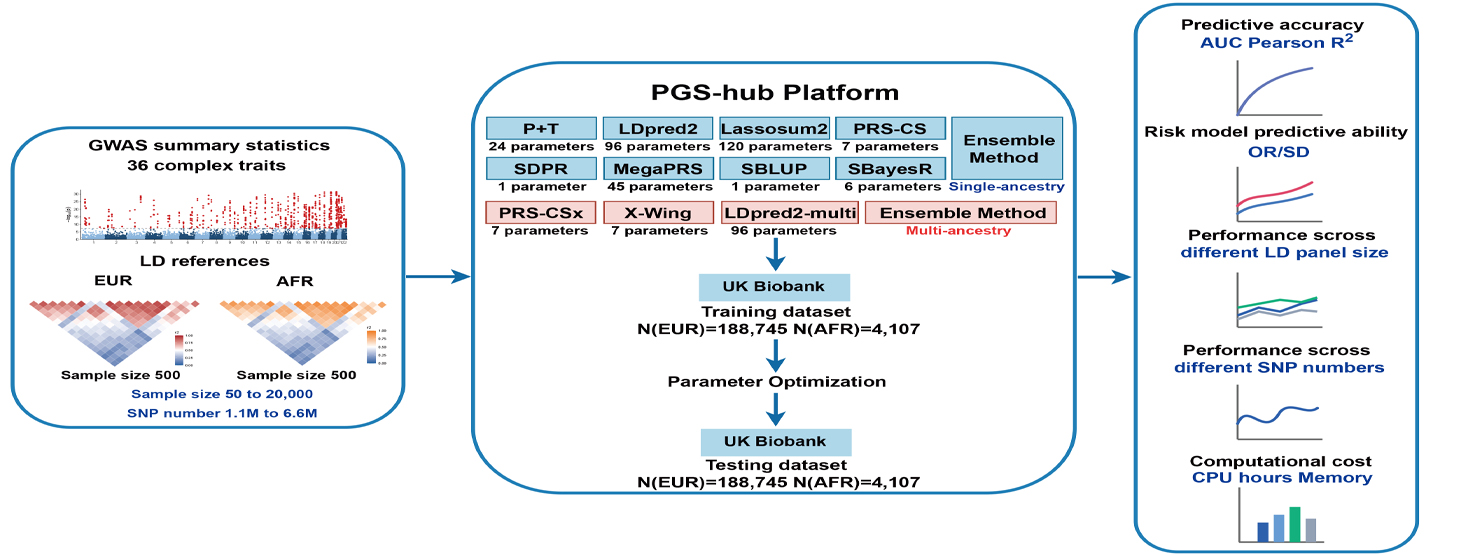
系统性评估单血统和多血统PGS方法概览






