国家生物信息中心揭示染色质高级结构在细胞核内定位的调控机制
染色质在细胞核内的三维空间分布对基因表达调控、DNA复制和DNA损伤修复等生命基本活动至关重要。然而,传统的Hi-C技术仅能测量染色质片段间的相互作用频率,无法直接提供染色质形成的高级结构在细胞核内的物理定位信息,限制了我们对染色质空间组织形式以及调控机制的深入理解。
8月29日,国家生物信息中心计算生物学部徐晨欢团队在Science Advances 期刊发表了题为“Architectural transcription factors collectively shape nuclear radial positioning of chromatin contacts”的研究论文。该研究开发了新型染色质构象捕获技术Radial-C,实现了对染色质相互作用沿细胞核径向轴(从核边缘到核中心)的精准定位,并揭示了不同转录因子可以“集体”调控染色质的径向分布。
研究团队利用微球菌核酸酶(MNase)在细胞核内的连续扩散特性,通过精确控制酶的扩散与消化时间(1、5、20分钟),构建了覆盖核边缘到核中心的染色质互作图谱。结合染色质互作频率,团队进一步构建了“径向得分”体系,将染色质高级结构定位到细胞核的径向轴上。研究发现,染色质高级结构沿细胞核径向轴呈现多样性:核外围区域富集沉默的异染色质结构,而核内部区域则富含活跃的染色质,如染色质环和核斑结构等。此外,研究还发现染色质环的挤压方向与其内部区域的径向定位存在关联。
染色体间相互作用(trans contacts)因为来源复杂,通常是Hi-C研究的“禁区”,生物学功能鲜有报道。研究团队凭借“径向得分”,成功筛选出由径向上相互邻近的染色质形成的染色体间相互作用,发现它们与染色体内相互作用(cis contacts)表现出几乎一致的活跃染色质/异染色质的区室化特征(A/B compartments),证明A/B区室分离是染色质的内在保守属性,与互作对象无关。
研究团队还通过急性干扰多种转录因子在染色质上的结合,发现染色质相互作用的减弱通常伴随着两个互作位点沿径向轴的分离。这暗示染色质相互作用不仅仅是两个互作位点在细胞核内相互邻近的结果,其本身也能作为作用力参与调控染色质在细胞核内的空间定位。
综上,本研究绘制了一套锚定细胞核物理坐标的染色质互作图谱,揭示了多个转录因子共同塑造染色质空间构象的调控机制,为理解发育、疾病中基因组结构的变化提供了新的研究工具和理论基础。
国家生物信息中心徐晨欢研究员为本文的通讯作者,博士研究生白娟、隗涵涵、已毕业博士生胡思玲为本文的共同第一作者。该研究得到了国家重点研发计划、国家自然科学基金、中国科学院先导专项等项目的资助。
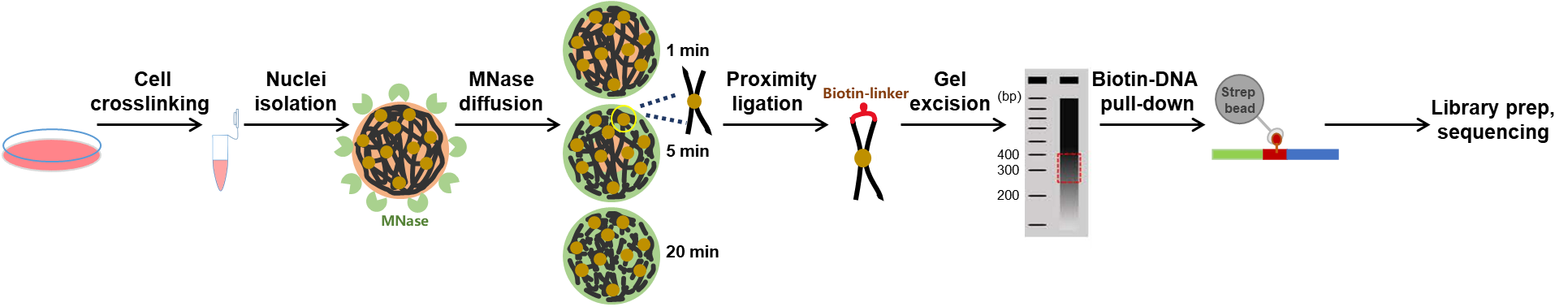
Radial-C方法的原理与基本流程






